Molekulare Diagnostik, Molekulare Therapie und Experimentelle Transplantation
Die Abteilung und Forschungsgruppe ist eine Diagnostik- und zugleich Forschungsabteilung mit der Zielsetzung, molekulare Diagnostik, molekulare Therapie und experimentelle Transplantation durch eine patientennahe Versorgung von „Bench-to-Bedside“ für eine patientennahe Versorgung weiter zu entwickeln. Die Abteilung bietet die molekulare Diagnostik aller bekannten angeborenen, primären Immundefekte, sowie die molekulare Diagnostik ausgewählter Hämatopoesedefekte an. Die Auftragsdiagnostik von Immun- und Hämatopoesedefekten wird erkrankten Patienten sowie deren Familienmitgliedern in Zusammenarbeit mit Kliniken, Ärzten und Genetikern bereitgestellt. Der Forschungsbereich fokussiert sich auf die Aufklärung von bisher nicht charakterisierten Entwicklungs- und Funktionsstörungen der lymphatischen, myeloiden und erythroiden Reihe, sowie von Autoimmunerkrankungen und Autoinflammationen.
Abteilungsleitung
Forschungsthemen
In mehr als 500 Genen des Menschen wurden bislang Varianten beschrieben, die zu angeborenen Störungen des Immunsystem („inborn Errors of Immunity“ IEI) führen können. Neben Defizienzen des Immunsystems können dabei sowohl Autoimmunitäten als auch Autoinflammationen Bestandteile der klinischen Erscheinungsbilder der Patienten sein. Die Diagnostik dieser Erkrankungen umfasst die genetische Analyse, bei der die Varianten in der genomischen DNA der Patienten aufgespürt werden, sowie die Aufklärung der pathomechanistischen Vorgänge, die zu den verschiedenen Erkrankungen beim Vorliegen der einzelnen Genvarianten führen.
Diese pathophysiologischen Analysen können bisweilen sehr aufwändig sein und hin zur Nutzung von Zellmodellen, wie den humanen induzierbare Stammzellen, in die, unter Verwendung des CRISPR/Cas9 -Systems, die einzelnen Varianten zu nachfolgenden experimentellen Analysen eingebracht werden können. Immer wieder können dabei bei Patienten neue Gendefekte identifiziert werden und aus den identifizierten Störungen der immunbiologischen Abläufe Erkenntnisse zur komplexen Funktionsweise eines intakten menschlichen Immunsystems gewonnen werden.
Die frühe Erkennung von Bakteriämie, die Identifikation des Pathogens sowie möglicher begleitender Pathogenitätsfaktoren und antimikrobieller Resistenzen sind von hoher klinischer Bedeutung. So sinkt beispielsweise im Kontext von Sepsis die Überlebensrate um circa 8% pro Stunde bei verzögerter Therapieeinleitung. Im transfusionsmedizinischen Kontext spielt die Pathogendetektion beispielsweise im Rahmen von Stammzelltransplantationen, oder in der Herstellung von Blutprodukten eine Rolle. Kontaminationen von Blutprodukten, insbesondere Thrombozytenkonzentraten mit Bakterien, Viren und anderen Pathogenen ist ein ernstes Problem, das sowohl die Qualität des Transfusionsprodukts als auch die Sicherheit des Empfängers beeinträchtigt.
Im Kontext von Sepsis spiegelt das Dilemma der verzögerten Diagnose von Infektionskrankheiten und insbesondere Sepsis sich in den aktuellen diagnostischen Kriterien für Sepsis wider, die eine vermutete oder bestätigte Infektion in Kombination mit einer Erhöhung von zwei Punkten im Sequential Organ Failure Assessment (SOFA) Score erfordern. Wie der Name schon sagt, steigt dieser Score, wenn ein Organversagen bereits erkennbar ist. Die Abteilung entwickelt zu dieser Problematik neue Methoden.
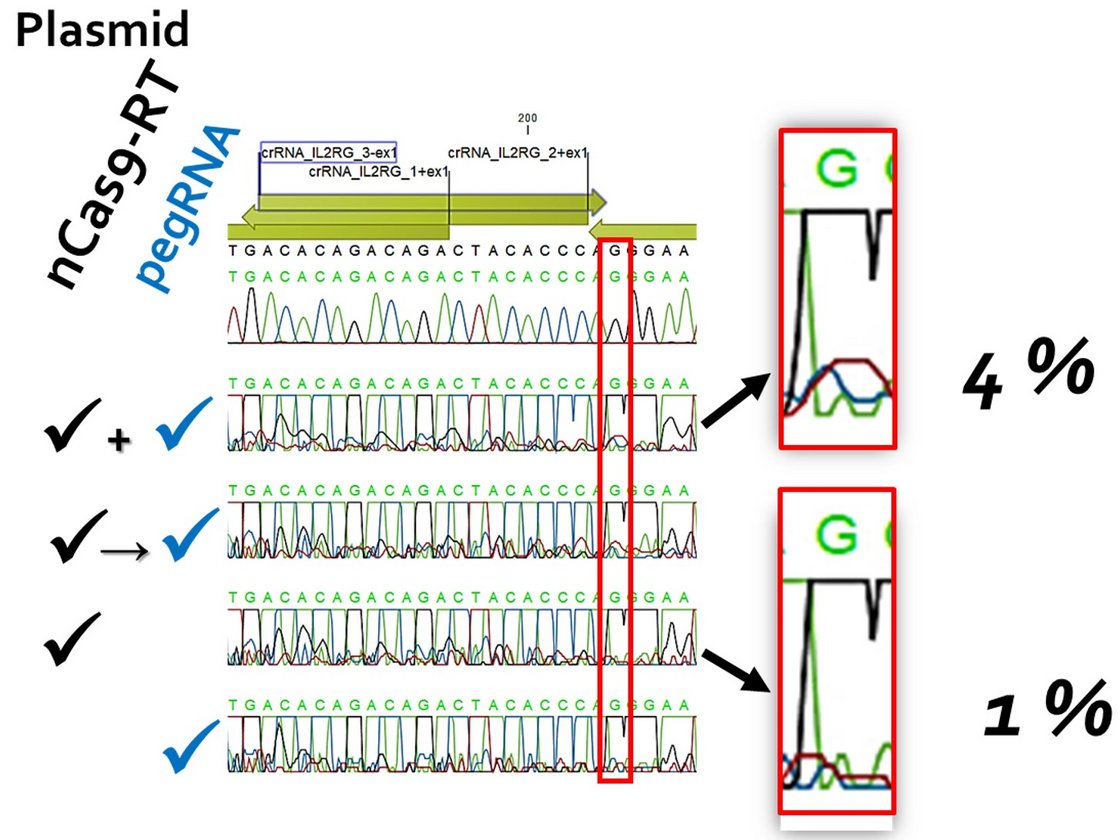
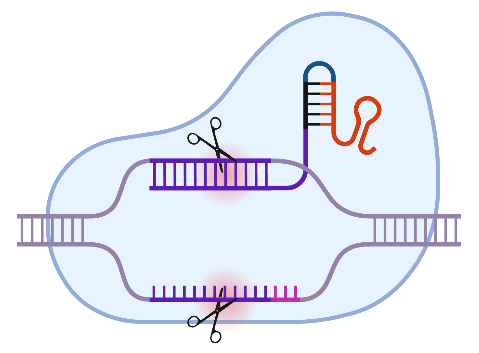
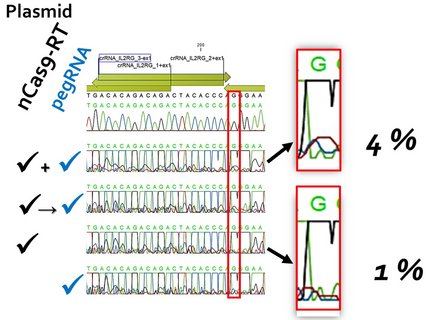
Es gibt schätzungsweise etwa 10 000 monogenetischen Erkrankungen des Menschen. Dazu zählt auch der X-chromosomale schwere kombinierte Immundefekt (X-SCID), der rund 50 % aller SCID-Fälle ausmacht. Er wird durch Mutationen im IL2RG-Gen verursacht. Nun hat durch die Verfügbarkeit des CRISPR/Cas-Systems die Erforschung nicht-viraler Genkorrektur-Ansätze für derartige Krankheiten einen bedeutenden Impuls erhalten. Dabei ist das Prime Editing (PE) eine vielversprechende Plattform, nicht zuletzt durch den Einsatz einer Cas9-Variante, die statt eines DNA-Doppelstrangbruchs einen Einzelstrangbruch ("nick") einführt. Weitere Verbesserungen der Methodik werden kontinuierlich vorgestellt. Diese betreffen sowohl das Design der PE-Komponenten wie auch die Manipulation des Target-Zell-Stoffwechsels, um beispielsweise unerwünschte Effekte des intrinsisch notwendigen "Mismatch"-Reparaturmechanismus auszuschalten.
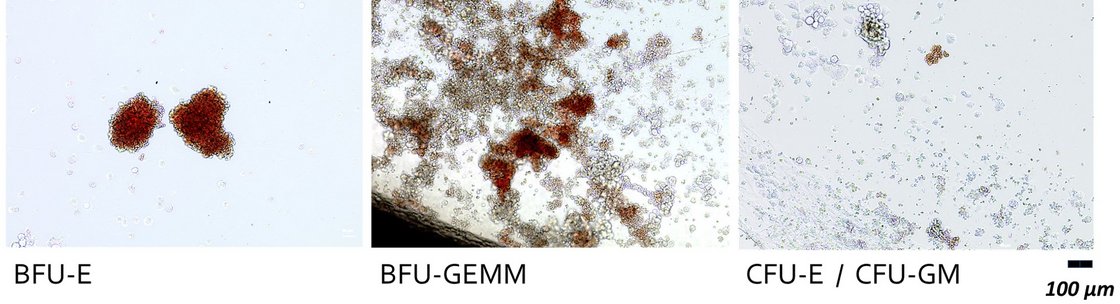
Das Wunschziel wäre, mit Effizienzen > 50 % Mutationen gezielt im IL2RG-Gen in humanen CD34+-Blutstammzellen ex vivo zu korrigieren. Dabei sollen die Zellen nach der Behandlung a) ihre Stammzell-Eigenschaften und b) ihre Fähigkeit für eine Langzeit-Repopulation behalten. Grundsätzlich möchten wir ein Protokoll definieren, das mit möglichst wenigen molekularen Werkzeugen auskommt. Die erste Aufgabe soll also sein, im vorgegebenen experimentellen Rahmen die Effizienz zu verbessern.
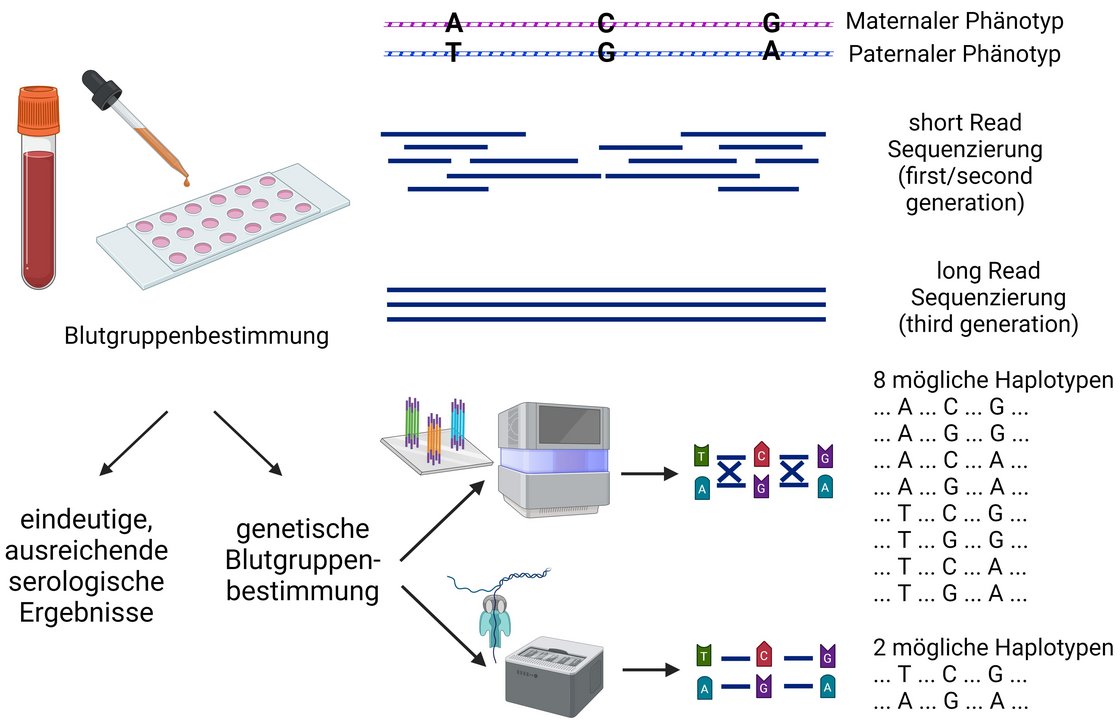
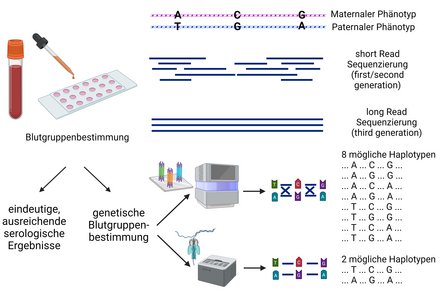
Third-Generation-Sequencing (TGS) bezieht sich auf eine neuartige Sequenzierungstechnologie, die DNA- oder RNA-Fragmente in ihrer ganzen Länge sequenzieren kann. Während TGS hinsichtlich Bearbeitungsdauer und Durchsatz verglichen mit konventioneller Second-Generation-Sequencing-Technologie Nachteile aufweist, so bietet TGS bezüglich Sequenzierung komplexer genetischer Fragestellungen Vorteile, da es sowohl eine skalierbare Technologie darstellt, als auch die Analyse langer DNA- oder RNA-Fragmente ermöglicht.
Long-Reads sowie weitere Vorteile ermöglichen einige Anwendungsgebiete mit Bezug zur Transfusionsmedizin. Aktuelle Anwendungsgebiete sind die Sequenzierung von mikrobiellen Pathogenen inklusive Virulenz- und Ressistenzbestimmung. Weiterhin wird die Technik zur Auflösung von manchen Genen wie dem NCF1-Gen angewandt, welches mit herkömmlicher Short-Read-Sequenzierung aufgrund der Problematik zweier Pseudogene nur erschwert untersucht werden kann. Dies sowie die Auflösung von Haplotypen ist auch ein Anwendungsgebiet in der Blutgruppendiagnostik, beispielsweise wie dargestellt im Rhesusblutgruppensystem.
Methodenspektrum
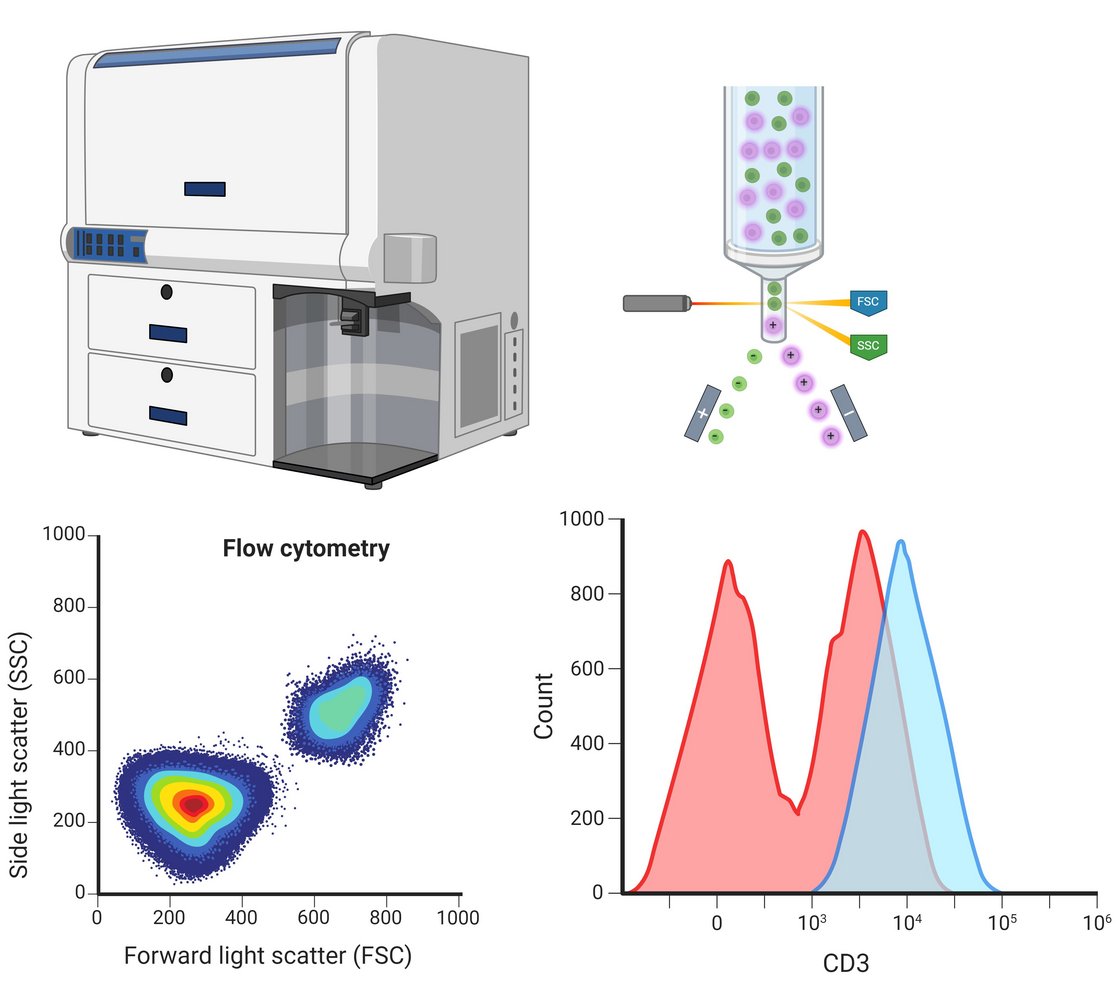
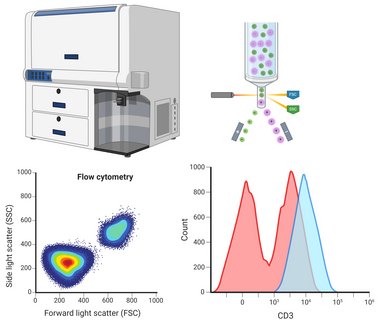
Die Methodik der Durchflusszytometrie wird in der Abteilung schwerpunktmäßig zur Analyse von Leukozyten, Thrombozyten und Stammzellen eingesetzt. Ein konkretes Anwendungsbeispiel ist die Aufklärung angeborener Fehler des Immunsystems, um hämatopoetische Zellen anhand von Oberflächenmarkern multiparametrisch quantitativ zu analysieren und Zielzellen per Sortierung aufzureinigen. Als Geräte stehen dabei vier Durchflusszytometer zur Verfügung: Zwei reine Analysegeräte mit zwei Lasern und fünf Auswertekanälen bzw. mit fünf Lasern und 30 Auswertekanälen sowie zwei Zellsortierer mit zwei Lasern und acht Auswertekanälen bzw. mit drei Lasern und 15 Auswertekanälen.
Im Rahmen der Forschungs- und Diagnostik-Arbeit kommt in unserer Abteilung ein breites Repertoire an molekulargenetischen und molekularbiologischen Methoden zum Einsatz. Eine zentrale Gruppe der molekulargenetischen und molekularbiologischen Methoden bildet die Arbeit mit Nukleinsäuren und deren Analyse. Das beinhaltet unter anderem die Polymerase-Kettenreaktion (PCR), die eine selektive Amplifikation von bestimmten Segmenten der DNA ermöglicht. In der Abteilung sind zahlreiche fortgeschrittene Varianten der PCR, wie die Reverse-Transkriptase quantitative PCR (RT-qPCR), welche eine Detektion und Quantifikation spezifischer RNA- Sequenzen ermöglicht, etabliert.
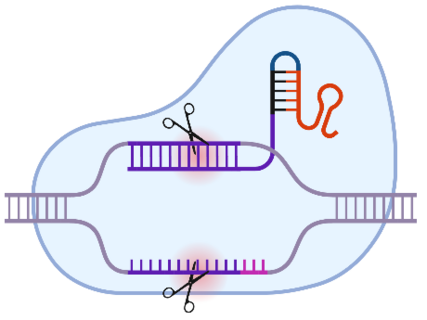
Ebenfalls ist ein mikrobiologisches Labor vorhanden, in dem zum Beispiel die rekombinante Expression von Proteinen aus gentechnisch modifizierten E. coli-Bakterien durchgeführt wird. Weitere Verfahren ermöglichen die Analyse der Expressionsprodukte: Western Blotting beispielsweise erlaubt die Identifizierung des Zielproteins in einem Proteingemisch auf Basis von dessen Größe und spezifischer fluoreszenzgekoppelter Antikörper gegen das Protein. Häufig in der Abteilung eingesetzt werden diese Verfahren für die Expression von rekombinanten Cas9-Proteinen. In Kombination mit einem zielspezifischen Leitmolekül, der guide-RNA (gRNA), lassen sich so gezielte Schnitte im Genom setzen. Mit weiterführenden Modifikationen der Technik sind sogar spezifische Modifikationen bestimmter Basen oder das Abfangen von gezielten DNA-Abschnitten aus dem Gesamtgenom möglich.
Auch für die Entwicklung und Durchführung zellbasierter Assays ist die Abteilung ausgestattet. Mehrere Zellkulturbänke und Inkubatoren ermöglichen eine sterile Kultivierung humaner Zellen. Für Folgeanalysen stehen beispielsweise verschiedene Mikroskope, unter anderem auch ein Laser-Scanning-Mikroskop bereit.
Das Vorhandensein dieser Vielzahl an etablierten Methoden und Geräten ermöglicht der Abteilung die Bearbeitung verschiedenster Fragestellungen, sowohl in Projekten der Routine-Diagnostik als auch im Forschungsbereich.
In unserer Abteilung kommen verschiedenste Sequenziertechniken zur Analyse von DNA- und RNA-Molekülen zum Einsatz. Generell werden durch durch eine durchdachte Mischung von Sequenziertechnologien jeder Generation Projekte je nach Anforderung ermöglicht und optimal bearbeitet.
Unsere Sequenzierplattformen umfassen:
- First Generation Sequencing: Die klassische Methode nach Sanger, die eine hohe Genauigkeit und Zuverlässigkeit bietet. Diese Methode eignet sich für die Sequenzierung von kurzen DNA-Abschnitten, wie z. B. einzelnen Genen oder Exonen. Wir verwenden ein modernes Kapillarelektrophorese-Gerät, das eine hohe Auflösung und Sensitivität ebenso, wie auch einen hohen Probendurchsatz von bis zu 96 Proben pro Stunde, gewährleistet.
- Second Generation Sequencing: Diese Methode mit extrem hoher Kapazität eignet sich durch massive Parallelisierung und eine hohe Sequenzierkapazität für die Sequenzierung extrem hoher Probenzahlen oder großer Bereiche des menschlichen Genoms. Vor allem die Sequenzierung großer Genpanels in diversen Patientenkohorten oder Zellpopulationen wird hier für verschiedenste Forschungsprojekte verwirklicht.
- Third Generation Sequencing: Mittels dieser Technik wird die Sequenzierung von einzelnen Nukleinsäure-Molekülen mittels sehr langer Leselängen ermöglicht. Es stehen in der Abteilung verschiedene Gerätemodelle zur Verfügung, die einen schnellen Wechsel bezüglich Kapazität, Dauer und Kosteneffizienz ermöglichen. Zum Einsatz kommt diese Technologie für Sequenzierungen und Analysen von komplexen oder repetitiven Genomregionen, großen strukturellen Veränderungen und Transkripten.

Das menschliche Immunsystem ist ein komplexes Netzwerk von Zellen und Organen, das eine entscheidende Rolle bei der Abwehr von Krankheiten spielt. Eines der zentralen Organe des Immunsystems ist der Thymus, der eine Schlüsselrolle bei der Ausbildung und Reifung von T-Zellen spielt. In den letzten Jahren hat die Forschung bedeutende Fortschritte bei der Entwicklung von "artifiziellen Thymus-Organoide" (ATO) gemacht. Diese innovative Technologie ermöglicht es uns die Störung der T-Zellentwicklung bei verschiedenen Gendefekten detailliert in den Fokus zu nehmen.
Die Thymus-Organoide setzen sich aus einer gentechnisch optimierten murinen Stromazelllinie sowie humanen hämatopoetischen Stammzellen zusammen. Diese CD34-positiven Stammzellen können entweder aus T-Zell-defizienten Patienten und Vergleichspersonen stammen oder wir können diese aus humanen induzierten pluripotenten Stammzellen (hIPSC) generieren, in die, per CRISPR/Cas-Technologie, genetische Varianten von Interesse eingebracht werden.
Die ATOs erlauben uns ein exaktes Monitoring der T-Zelldifferenzierung über eine Reihe von definierten Progenitorstufen. Die Detektion und Zuordnung dieser verschiedenen Vorläuferpopulationen erfolgt in unserem Labor per Durchflusszytometrie. Auf diese Weise können wir Blockaden in der Entwicklung beim Vorliegen der verschiedenen Gendefekten sehr präzise einzelnen T-Zell-Differenzierungsstufen zuordnen.
Auffällige Zellpopulationen können wir dann per durchflusszytometrie-basierter Zellsortierung isolieren und im Anschluss umfassend genetisch (z.B. Genexpressionsanalysen per RNASeq) sowie zell- und molekularbiologisch (z.B. Bestimmung von Oberflächenmarker, Aktivierungsassays, Signaltransduktions-analysen) charakterisieren um die, den einzelnen angeborenen Störungen des T-zellulären Immunsystems zugrundeliegenden, Pathomechanismen umfassend zu analysieren und aufzuklären.
Lehre und Abschlussarbeiten
Wir bieten regelmäßig Praxissemester, Bachelorarbeiten, Masterarbeiten und Doktorarbeiten an. Die Abschlussarbeiten werden eng betreut durch die Wissenschaftler-/innen, das technische Personal und die Abteilungsleitung verbunden mit einem Einblick in unser breites Methodenspektrum. Aufgrund der komplexen Themen ist im Regelfall ein längeres Engagement (9 Monate, in Einzelfällen 3-6 Monate) erforderlich. Bei Interesse bewerben Sie sich bitte mit Anschreiben, Lebenslauf und bisherigen Noten bei der Abteilungsleitung. Darüber hinaus engagiert sich die Abteilung in der Lehre des Instituts für Transfusionsmedizin sowie in der AG Lehrforschung der Medizinischen Fakultät der Universität Ulm.
Routinediagnostik der Abteilung (IKT)
Kooperationen
- BioLAGO e.V. – The health network
- Dr. D. Jovanovski, Prof. Dr. A. Liebold (Department of Cardiothoracic and Vascular Surgery, University Medical Center Ulm)
- Prof. Dr. B. Nilsson (Department of Immunology, Genetics and Pathology, University Uppsala, Schweden)
- Prof. Dr. K. Nilsson Ekdahl (Department of Immunology, Genetics and Pathology, University Uppsala, Linnæus Center of Biomaterials Chemistry, Schweden)
- Prof. Dr. C. Schmidt (Institute of Pharmacology of Natural Products and Clinical Pharmacology, Ulm University)
- Prof. Dr. M. Weiß, Prof. Dr. E. Barth (Department of Anaesthesiology and Intensive Care Medicine, University Medical Center Ulm)
- Prof. Dr. S. Ehl (Centre for Chronic Immunodeficiency, University Medical Center Freiburg)
- L. Wohlgemuth, Prof. Dr. Markus Huber-Lang (Institute for Trauma-Immunologie, University Medical Center Ulm)
Forschungsförderung
- Else Kröner-Fresenius-Stiftung
- DRK-Blutspendedienst Baden-Württemberg – Hessen